Platforma interaktywnej analizy danych (ang. interactive data science) wyposażona w system Jupyter (IDE przez przeglądarkę) pozwalająca na zgłaszanie zadań Apache Spark w językach Scala, Python, Julia oraz R.
Usługa jest przeznaczona dla naukowców, studentów, przedsiębiorców i innych osób zainteresowanych interaktywną analizą danych. Dostęp do usługi wymaga wypełnienia wniosku o grant obliczeniowy poprzez formularz. Więcej informacji na stronie głównej.
Po przyznaniu dostępu należy zapoznać się ze znajdującą się poniżej instrukcją użytkowania usługi.
Spis treści:
- Instrukcja dostępu do platformy Jupyter
- Uruchamianie notatek
- Zmiana jądra w notatkach
- Przykładowe notatki
- Dodawanie plików
- Pobieranie danych z Mostu Wiedzy
- Instalacja dodatkowych modułów Python
- Instalacja dodatkowych modułów przy użyciu Conda
- Użycie Julia w notatniku
- Instalacja dodatkowych modułów Julia
- Restart Jupyter
- Wprowadzenie do Apache Spark
- Użycie Apache Spark i Scala z notatnika
- Użycie Apache Spark i Python z notatnika
- Użycie Dask Gateway i Python z notatnika
- Użycie Apache Spark z konsoli
- Elementy uczenia maszynowego
- Rozpraszanie obliczeń w klastrze
- Generowanie wykresów przy użyciu plotly
- Instalacja scikit
- Biblioteka Tensorflow
- Tworzenie diagramów - DrawIO
- Zainstalowane biblioteki (Python3 base)
- Pomoc
Instrukcja dostępu do platformy Jupyter
Informacje dotyczące procesu otrzymania dostępu znajdują się na stronie głównej. W celu utworzenia konta użytkownika należy przejść do portalu SSO CI TASK pod adresem: https://sso.task.gda.pl i nacisnąć przycisk „Rejestracja”.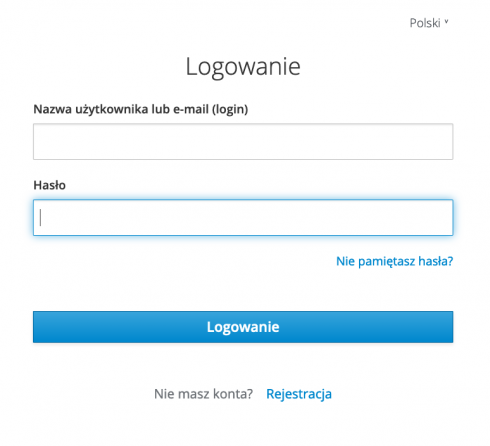
Następnie należy wprowadzić dane dla nowo tworzonego użytkownika. Obecnie możliwe jest utworzenie konta tylko dla osób posiadających adres email w jednej z domen Związku Uczelni w Gdańsku im. Daniela Fahrenheita (PG, UG i GUMed) (np. user@pg.edu.pl).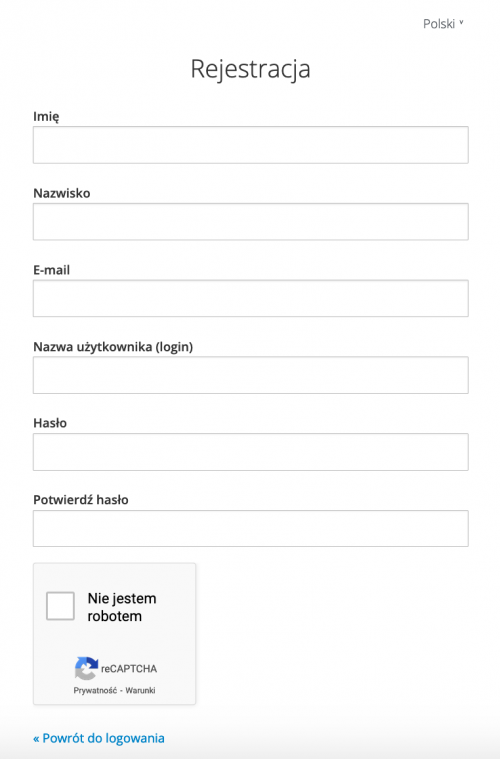
Następnie należy wypełnić wniosek o otrzymanie grantu obliczeniowego.
Informacje o przyznaniu dostępu zostanie przesłana pocztą elektroniczną. Po otrzymaniu dostępu do Jupyter można rozpocząć pracę z systemem pod adresem: https://jupyter.bigdata.task.gda.pl/
UWAGA: czas uruchomienia przy pierwszym logowaniu może wynieść nawet 10 minut.
Jeśli użytkownik nie jest aktualnie zalogowany pojawi się przycisk „Sign in with keycloak”. Należy go wybrać.
![]()
Nastąpi przekierowanie do portalu SSO CI TASK. Należy wprowadzić login i hasło wcześniej utworzonego użytkownika. Po poprawnym zalogowaniu się można przystąpić do pracy z Jupyter.
Z poziomu aplikacji Jupyter użytkownik jest widziany jako użytkownik systemowy jovyan.
Pliki zapisane w katalogu domowym (/home/jovyan) oraz jego podkatalogach są zapisywane w pamięci trwałej i są dostępne pomiędzy poszczególnymi uruchomieniami Jupyter. Pliki zapisywane w innych miejscach zostaną utracone po wylogowaniu się. Maksymalny rozmiar danych przechowywanych w katalogu domowym nie może przekroczyć 20GB. Użytkownik ma dostęp tylko do swoich danych tj. nie może obejrzeć katalogu domowego innych użytkowników.
Po zakończeniu pracy należy wylogować się z aplikacji Jupyter poprzez wybranie z górnego menu: File -> Log Out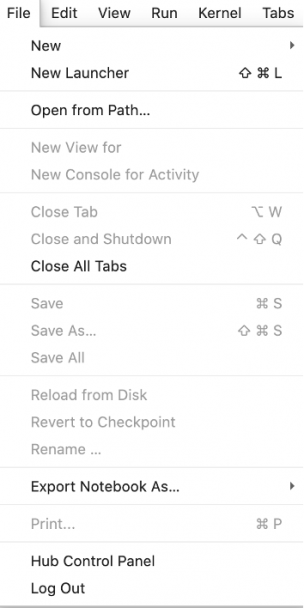
Uruchamianie notatek
W panelu po lewej stronie wyświetlana jest lista plików znajdujących się w katalogu domowym użytkownika:
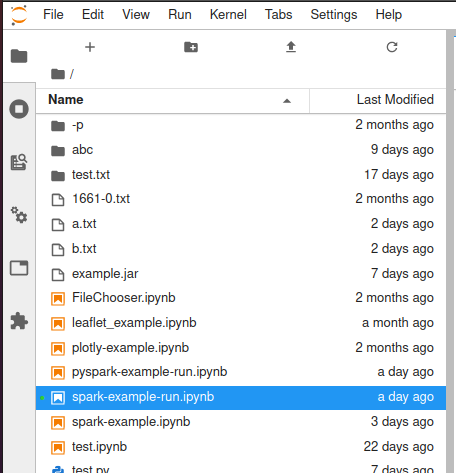
Pliki notatek są oznaczone pomarańczową ikoną oraz mają rozszerzenie .ipynb. Po wybraniu notatki, w oknie głównym ukaże się podgląd pliku:
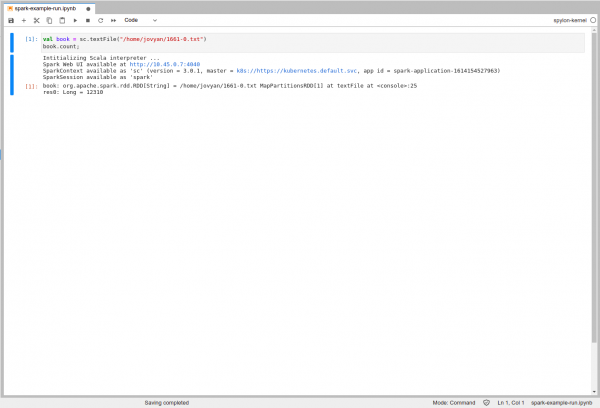
Po naciśnięciu ikony Run na dowolnej komórce wykonane zostaną zawarte w niej polecenia.

Zmiana jądra w notatkach
Po wybraniu notatnika wraz z opcjami operacji jakie możemy zastosować na notatniku, w prawym górnym rogu znajduje się nazwa jądra, z którego dany notatnik korzysta:

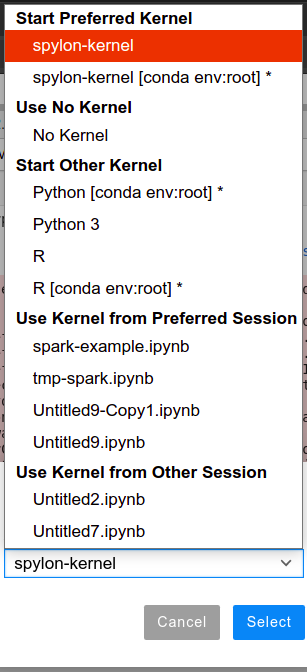
Jądro można zmienić w dowolnym momencie, na inne dostępne w środowisku wykonawczym poprzez wybranie z górnego menu: Kernel -> Change Kernel:
Przykładowe notatki
W celu zademonstrowania możliwości platformy przygotowaliśmy dla Państwa szereg przykładowych notatek. Notatki można obejrzeć na stronie projektu w GitLab.
W celu pobrania notatek do swojego katalogu domowego należy z górnego menu wybrać (File -> New -> Terminal)
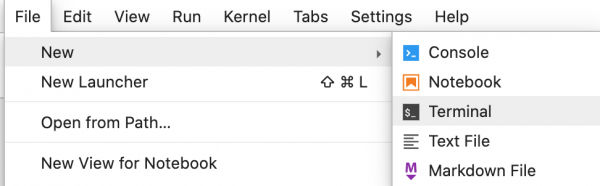
I następnie wykonać poniższe polecenie:
git clone https://projects.task.gda.pl/wojartic/most-danych-przyklady.git
Dodawanie plików
Pliki o rozmiarze mniejszym niż 20 MB mogą zostać pobrane z komputera lokalnego poprzez użycie górnego menu (ikona upload) aplikacji Jupyter. Nie jest możliwe lokalne pobranie plików o większym rozmiarze.
Do pobrania plików o rozmiarze większym niż 20MB należy umieścić plik na serwerze i skopiować jego adres publiczny (np. przy użyciu platformy Dropbox). Następnie w aplikacji Jupyter uruchomić nowy terminal poprzez File -> New -> Terminal.
W Terminalu trzeba pobrać plik używając polecenia wget, przykładowo:
wget https://adres_serwera/moj_plik
Jeśli potrzebujesz przechować dane o większym rozmiarze (np. własne repozytorium danych) napisz do nas.
Pobieranie danych z Mostu Wiedzy
Na stronie Mostu Wiedzy przejść do zakładki o nazwie Dane Badawcze:
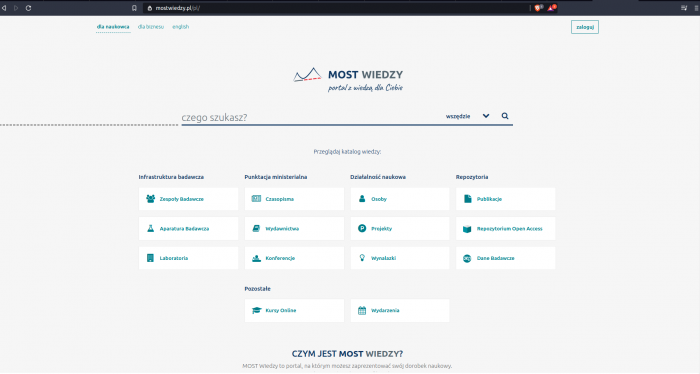
Po wejściu na nią, otworzy się wyszukiwarka z publikacjami naukowymi i zbiorami danych do pobrania:
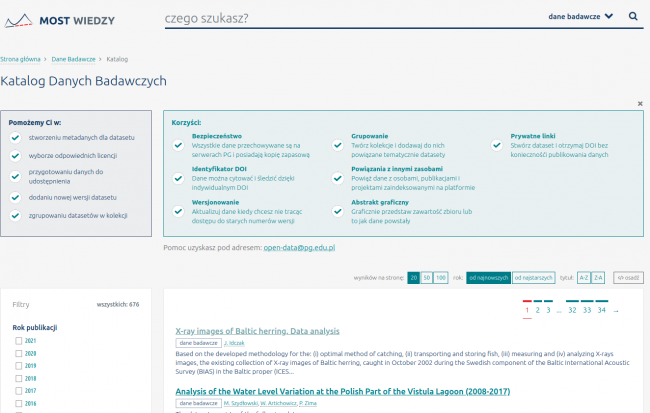
Po wejściu w żądane źródło ukazuję się dokument, na którym widnieje sekcja Plik z danymi badawczymi:
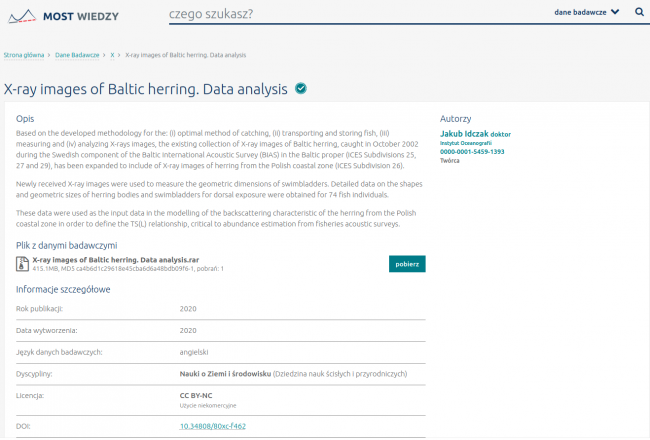
Celem pobrania zbioru danych, należy nacisnąć prawy przycisk myszy na przycisku pobierz:
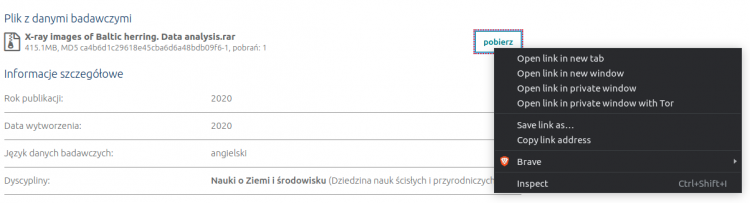
a następie wybrać opcję odpowiedzialną za kopiowanie adresu linku.
Mając skopiowany adres, należy w terminalu wpisać komendę:
wget --content-disposition https://mostwiedzy.pl/
,gdzie hiperłącze https://mostwiedzy.pl/ można wkleić za pośrednictwem skrótu klawiszowego Ctrl+V.
Instalacja dodatkowych modułów Python
Tylko dane zapisane w katalogu domowym użytkownika są dostępne pomiędzy poszczególnymi uruchomieniami Jupyterlab. Z tego też powodu nowe moduły muszą być instalowane lokalnie poprzez polecenie pip z użyciem opcji --user.
pip install --user nazwa_modulu
Lokalizację w ten sposób zainstalowanych modułów można sprawdzić poleceniem:
python -m site --user-site
Notatki Jupyter powinny używać kernela "Python 3". Dostęp do nowo zainstalowanych modułów może wymagać restartu kernela. Z górnego menu wybrać: Kernel -> Restart Kernel.
Instalacja dodatkowych modułów przy użyciu Conda
Conda to menadżer pakietów, środowisk i zależności, który jest najczęściej używany razem z językiem Python. W katalogu domowym użytkownika znajduje się plik .condarc, który informuje Conda, by nowe środowiska wirtualne były instalowane w katalogu /home/jovyan/user-environments/. Dzięki temu, moduły instalowane w środowiskach wirtualnych zostaną zapisane w pamięci trwałej i będą dostępne pomiędzy poszczególnymi logowaniami do Jupyter.
Uwaga: stworzenie nowego środowiska wirtualnego uniemożliwia dostęp do modułów zainstalowanych w środowisku głównym Conda. Oznacza to, że wszystkie moduły będą musiały być zainstalowane ponownie przez użytkownika (np. przy pomocy opcji --clone) w jego katalogu domowym. Z tego też powodu zachęcamy do instalacji wszystkich pakietów przy użyciu pip.
Najpierw należy uruchomić nowy terminal: File -> New -> Terminal.

W następnym kroku tworzymy środowisko wirtualne Conda. Nazwa środowiska jest dowolna, w tym przypadku będzie to "myenv":
conda create --yes --name myenv ipykernel
Jeśli chcemy, aby nasze środowisko posiadało dostęp do wszystkich aktualnie zainstalowanych modułów można sklonować główne środowisko condy:
conda create --yes --name myenv --clone base
Uwaga: klonowanie środowiska może trwać nawet kilkadziesiąt minut i zajmie ponad 2,5 GB w katalogu domowym użytkownika!
Po utworzeniu środowiska w menu wyboru plików po lewej stronie można zaobserwować pojawienie się w/w katalogu user-environments.
moduły można zainstalować w nowym środowisku poleceniem (module-name zamieniamy na nazwę modułu):
conda install --yes --name myenv module-name
na samym końcu należy aktywować środowisko:
conda activate myenv
Nowo utworzone środowisko można wykorzystać do uruchamiania notatek Jupyter. Należy utworzyć nową notatkę (File -> New -> Notebook) lub przejść do już istniejącej notatki i z górnego menu wybrać: Kernel -> Change Kernel:
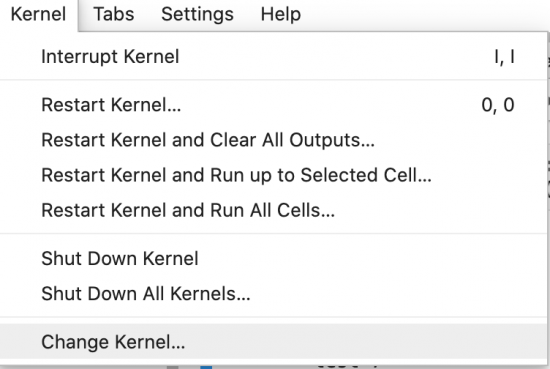
W nowym oknie z listy rozwijanej wybrać przed chwilą stworzone środowisko wirtualne myenv ([conda:myenv]) i zatwierdzić wybór przyciskiem Select.
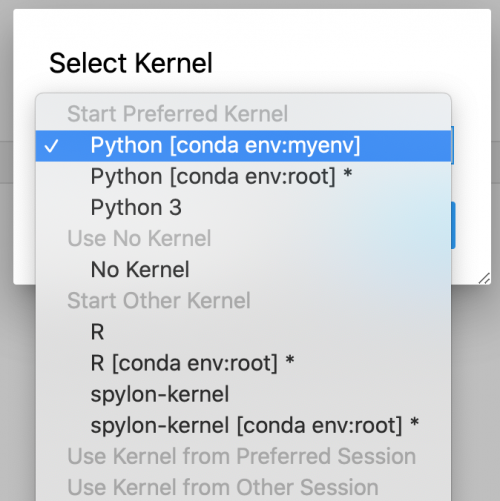
Poprawnie przeprowadzoną zmianę kernela można zaobserwować w prawym górnym rogu:

Jeśli utworzone przez nas środowisko nie jest widoczne w menu zmiany kernela, konieczne jest jednorazowe wykonanie restartu kontenera według informacji zawartych w sekcji: Restart Jupyter.
Uwagi
Środowisko można usunąć poleceniem:
conda remove --name myenv --all
Jeśli środowisko było wcześniej używane przez notatki, przed usunięciem należy przejść do odpowiedniej notatki i z górnego menu wybrać: Kernel-> Shut Down Kernel.
Plik konfiguracyjny .condarc jest kopiowany do folderu użytkownika przy starcie kontenera pod warunkiem, że plik ten do tej pory nie istniał. Z tego względu możliwe jest wprowadzenie własnych zmian w tym pliku. W razie potrzeby odtworzenia podstawowej konfiguracji należy usunąć / przenieść plik .condarc i następnie zrestartować serwer:
rm /home/jovyan/.condarc
Więcej informacji na temat stosowania Conda można znaleść w dokumentacji.
Użycie Julia w notatniku
W aplikacji Jupyter możliwe jest tworzenie notatników przy użycia języka Julia w wersji 1.6.3. W celu wybrania odpowiedniego dla Julii kernela należy kliknąć w prawym górnym rogu notatki na nazwę kernela Julia 1.6.3:
![]()
Z podręcznego menu należy wybrać tę opcję, która odnosi się do języka Julia.
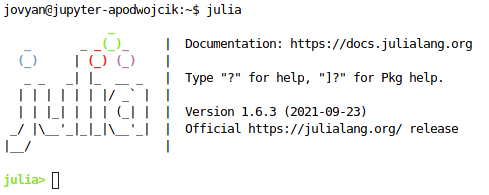
W przypadku używania Julii z poziomu terminala wystarczy, przed zastosowaniem składni języka, wykonać polecenie julia, tak jak na poniższym rysunku.
Instalacja dodatkowych modułów Julia
W ramach pakietu podstawowego zostały zainstalowane takie biblioteki jak:
- CSV
- Combinatorics
- DataFrames
- Distributions
- GenericLinearAlgebra
- HDF5
- IJulia
- JLD
- MultivariateStats
- Plotly
- Plots
- PyCall
- PyPlot
- StatsBase
Jeśli zaszłaby potrzeba użycia poleceń, które nie są zawarte w bibliotekach pakietu podstawowego, istnieje możliwość instalacji przez użytkownika dodatkowych bibliotek. W tym celu należy posłużyć się poleceniami:
using Pkg
Pkg.add(„nazwa biblioteki”)
Instalacji można dokonać z poziomu terminala lub notatki Jupyter z ustawionym dla języka Julia kernelem. W celu sprawdzenia jakie biblioteki są dostępne z poziomu użytkownika należy posłużyć się poleceniem:
Pkg.status()
Restart Jupyter
W niektórych sytuacjach może okazać się konieczny restart bieżącego kontenera Jupyter. W tym celu należy z górnego menu wybrać: File -> Hub Control Panel.
Następnie należy kliknąć czerwony przycisk Stop My Server.

Po zatrzymaniu serwera pojawi się przycisk Start My Server. Należy go wcisnąć.

Następnie wcisnąć przycisk Launch Server. Jupyter zostanie zrestartowany a użytkownik zostanie przekierowany do nowego okna Jupyter-lab.
Wprowadzenie do Apache Spark
Platforma Jupyter oferuje możliwość uruchamiania aplikacji Apache Spark (3.0.2) w oparciu o Java 11 (11.0.9.1).
Spark to platforma obliczeniowa ogólnego przeznaczenia, złożona z wielu środowisk narzędziowych, zoptymalizowanych pod kątem różnorodnych aplikacji.
Uwaga: zasoby dostępne do przeprowadzania obliczeń rozproszonych są dzielone pomiędzy użytkownikami. Pamiętaj o jak najszybszym zwróceniu zasobów poprzez zatrzymanie jądra (Kernel -> Shut Down Kernel) lub zakończenie sesji Spark (sc.stop())
Użycie Apache Spark i Scala z notatnika
W celu możliwości uruchomienia notatek należy zmienić jądro na spylon-kernel lub spylon-kernel [conda env:root] * , jeżeli jest inne niż w obecnej notatce.
Wraz z pierwszym uruchomieniem inicjalizowana jest sesja spark-shell na podstawie domyślnego kontekstu, która zwraca wynik operacji w tej samej komórce. Domyślny kontekst tworzony jest automatycznie przez kernel Spylon.
Przykładowo, poniższa notatka przedstawia wynik operacji na rozproszonej kolekcji:
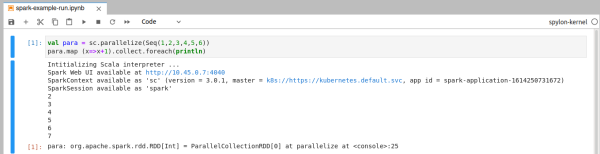
Widoczne hiperłącza nie są dostępne dla użytkownika, a po zakończeniu sesji danego notatnika, trwające obliczenia są kasowane.
Parametry sesji można zmienić poprzez stworzenie komórki zawierającej sekcję init_spark:
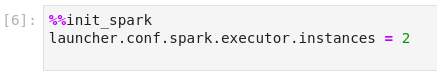
Domyślnymi parametrami konfiguracyjnymi są:
- Liczba instancji generowanych przez egzekutory Sparka:
launcher.conf.spark.executor.instances = 1
- Ilość pamięci do wykorzystania na proces egzekutora:
launcher.conf.spark.executor.memory = "1g"
- Liczba rdzeni do użycia w każdym module egzekutora.
launcher.conf.spark.executor.cores = 1
Pełna lista parametrów konfiguracyjnych znajduje się w oficjalnej dokumentacji Spark.
Uwaga: W jednym notatniku może być uruchomiona tylko jedna sesja SparkContext. Zatem zmiana parametrów konfiguracyjnych po inicjalizacji kontekstu i bez jego zatrzymania może wymagać restartu jądra (opcja: "Restart Kernel").
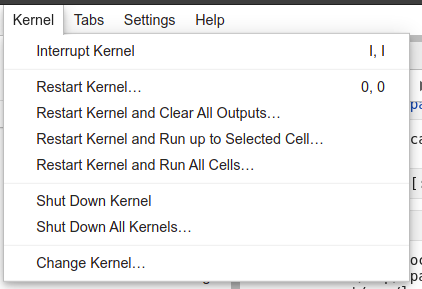
Użycie Apache Spark i Python z notatnika
W celu możliwości uruchomienia notatek należy zmienić jądro na Python 3 lub Python [conda env:root] * , jeżeli jest inne niż w obecnej notatce.
Zgłaszanie zadań dla Apache Spark jest możliwe tylko dla Python 3.8, który jest domyślną wersją języka zarówno dla jądra Python 3 jak i dla jądra Python 3 ze środowiskiem conda. Uprzednio wspomniane jądra zawierają biblioteki Numpy, Scipy oraz OpenCV. Niezbędne jest zainicjowanie SparkContext w notatniku przez użytkownika.
Domyślna inicjalizacja SparkContext odbywa się w następujący sposób:
import pyspark
sc = pyspark.SparkContext()
Przykładowo na podstawie notatnika pyspark-example-run.ipynb można utworzyć kolekcję równoległą:

Po zakończeniu sesji danego notatnika, trwające obliczenia są kasowane.
Parametry konfiguracyjne w kontekście możemy zmieniać za pomocą klasy konfiguracyjnej:

Domyślne parametry to:
- Liczba instancji generowanych przez egzekutory Sparka:
conf.set("spark.executor.instances", 1)
- Ilość pamięci do wykorzystania na proces egzekutora:
conf.set("spark.executor.memory", "1g")
- Liczba rdzeni do użycia w każdym module egzekutora.
conf.set("spark.executor.cores", 1)
Pełna lista parametrów znajduję się w oficjalnej dokumentacji Sparka.
Uwaga: W jednym notatniku może być uruchomiona tylko jedna sesja SparkContext. Zatem zmiana parametrów konfiguracyjnych po inicjalizacji kontekstu i bez jego zatrzymania może wymagać restartu jądra.

Użycie Dask Gateway i Python z notatnika
W celu możliwości uruchomienia notatek należy zmienić jądro na Python 3 lub Python [conda env:root] * , jeżeli jest inne niż w obecnej notatce.
Zgłaszanie zadań dla Dask Gateway jest możliwe tylko dla języka programowania Python, który jest domyślną wersją języka zarówno dla jądra Python 3 jak i dla jądra Python 3 ze środowiskiem conda. Wszystkie moduły na bazie których działa Dask Gateway, zostały uprzednio zainstalowane. W celu przeprowadzania obliczeń na klastrze Dask Gateway niezbędne jest zainicjalizowanie połączenia z API Dask Gateway poprzez jej odpowiedni moduł:
import dask_gateway
gateway = dask_gateway.Gateway()
Następnie należy zdefiniować zasoby jakie mają być dostępne do użytku przez jednego Workera oraz obraz z jakiego ma być stworzony. W przypadku obrazów z prywatnego rejestru, należy wskazać nazwę sekretu służącego do uwierzytelnienia użytkownika korzystającego z platformy. Zaleca się pozostawienie domyślnej nazwy obrazu aby uniknąć błędów wynikających z różnych wersji oprogramowania między Dask-Gateway a Schedulerem i Workerami.
Po wyspecyfikowaniu ustawień, można założyć nowy klaster obliczeniowy składający się z Dask Schedulera w przestrzeni nazw użytkownika. Liczbę Workerów można sterować naciskając odpowiednie strzałki na wygenerowanym panelu:
Lub z poziomu funkcji:
- dla skalowania statycznego, przykładowo:
cluster.scale(2)
- dla skalowania adaptacyjnego, przykładowo:
cluster.adapt(minimum=1, maximum=2)
Do zgłaszania zadań na uprzenio utworzonym klastrze obliczeniowym, służy połączenie klienckie:
W wygenerowanym oknie pod komórką jest link do interfesju, pomocnego do eksploracji wykonywanych obliczeń na klastrze. Z powodów bezpieczeństwa, został on zablokowany i aby się do niego dostać należy wkleić go do polecenia w terminalu na komputerze lokalnym użytkownika:
kubectl port-forward svc/ 8787:8787 --namespace <nazwa-użytkownika>
Oraz w nowym oknie przeglądarki wpisać url:
localhost:8787
Gdzie nazwę serwisu można znaleźć w przestrzeni nazw użytkownika, za pomocą polecenia:
kubectl get all --namespace <nazwa-użytkownika>
Tak zdefiniowane środowisko jest już w pełni skonfigurowane do przetwarzania zadań. Przykładowo, można wygenerować zestaw danych:
Który następnie należy zapisać w pamięci RAM Workerów:
Oraz użyć klasteryzacji w celu zbadania centroidów zbiorów danych:
Obliczenia powinny się pojawić na panelu wizualizacyjnym w przeglądarce. Po zakończonym zadaniu można wyświetlić rezultaty korzystając z biblioteki matplotlib.
Scheduler i Workery w przestrzeni nazw użytkownika wygasają po 15 minutach bezczynności.
Użycie Apache Spark z konsoli
Poza pisaniem notatek można wykorzystać standardowe narzędzia Apache Spark poprzez wywołanie poleceń spark-shell, spark-submit i pyspark dostępnych po ścieżką $SPARK_HOME/bin
spark-shell
Powłoka Sparka jest interfejsem do interaktywnej analizy danych.
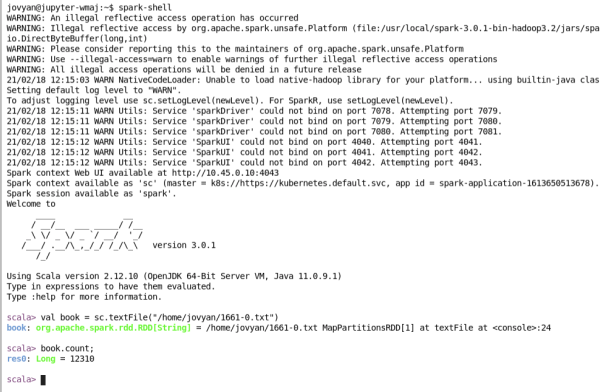
Możemy zmieniać domyślną konfigurację dodając parametry po dwóch myślnikach w taki sposób:
spark-shell --executor-cores 4
Pełna lista parametrów ukazuje się używając dyrektywy:
spark-shell --help
spark-submit
${SPARK_HOME}/bin/spark-submit to polecenie Spark używane do uruchamiania aplikacji w klastrze. Na przykładzie plików zawartych w katalogu ${SPARK_HOME}/examples możemy przygotować:
Wykonywanie plików java archive na przykładzie spark-examples_2.12-3.1.2.jar:
${SPARK_HOME}/bin/spark-submit --class org.apache.spark.examples.SparkPi ${SPARK_HOME}/examples/jars/spark-examples_2.12-3.1.2.jar
Wykonywanie plików z rozszerzeniem .py na przykładzie pi.py:
${SPARK_HOME}/bin/spark-submit ${SPARK_HOME}/examples/src/main/python/pi.py
W przypadku przytłaczającej liczby logów z aplikacji należy dodać 2>/dev/null na końcu polecenia w sposób:
${SPARK_HOME}/bin/spark-submit ${SPARK_HOME}/examples/src/main/python/pi.py 2>/dev/null
Można wyświetlić listę dostępnych opcji konfiguracyjnych za pomocą:
${SPARK_HOME}/bin/spark-submit --help
lub w oficjalnej dokumentacji Sparka.
pyspark
Interfejs Python dla aplikacji Spark.
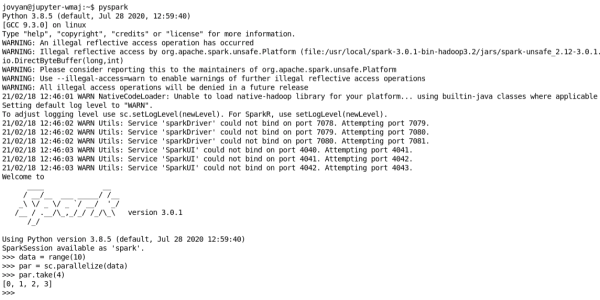
Możemy zmieniać domyślną konfigurację dodając parametry po dwóch myślnikach:
pyspark --executor-cores 4
I wyświetlić jakie parametry mamy do dyspozycji za pomocą:
pyspark --help
Więcej informacji na temat parametrów znajduję się w oficjalnej dokumentacji Spark Python API.
Elementy uczenia maszynowego z PyTorch
PyTorch jest platforma uczenia maszynowego typu open source, mocno znintegrowaną z komponentami niskopoziomowymi.
Można definiować sieci głebokiego uczenia w sposób klasowy:
class NeuralNetwork(nn.Module):
def __init__(self):
super(NeuralNetwork, self).__init__()
self.flatten = nn.Flatten()
self.linear_relu_stack = nn.Sequential(
nn.Linear(28*28, 512),
nn.ReLU(),
nn.Linear(512, 512),
nn.ReLU(),
nn.Linear(512, 10),
)
def forward(self, x):
x = self.flatten(x)
logits = self.linear_relu_stack(x)
return logits
Domyślnym nośnikiem obliczeń jest CPU, w celu zmiany na GPU należy zmienić jednostkę przetwarzającą:
device = 'cuda' if torch.cuda.is_available() else 'cpu'
Po wygenerowaniu przykładowych danych należy je nanieść na model, co pozwoli uzyskać predykcje:
X = torch.rand(1, 28, 28, device=device)
logits = model(X)
pred_probab = nn.Softmax(dim=1)(logits)
y_pred = pred_probab.argmax(1)
print(f"Predicted class: {y_pred}")
Rozpraszanie obliczeń w klastrze
Spark natywnie wspiera wbudowaną bibliotekę mllib. Definicje modeli uczenia maszynowego są już w niej zaimplementowane:
from pyspark.mllib.classification import SVMWithSGD
sc = SparkContext(...)
model = SVMWithSGD.train(data=..., iterations=...)
W przypadku uczenia głębokiego dla PyTorch przy użyciu rdzeni wykonawczych Spark, należy skorzystać z rozproszonej platformy Horovod. W tym celu należy:
- Zainicjalizować Sesję Spark oraz miejsce przechowywania zasobów:
spark = pyspark.sql.SparkSession.builder.getOrCreate()
store = horovod.spark.common.store.Store.create('.')
- Stworzenie tła obliczeniowego
backend = horovod.spark.common.backend.SparkBackend(num_proc=1)
- Stworzenia estymatora do uczenia
torch_estimator = horovod.spark.torch.TorchEstimator(backend=backend, store=store, model=...)
W estymatorze należy podać zainicjalizowany backend, store, stworzony model uczenia maszynowego oraz dodatkowe parametry, które należy podać dla konkretnego przypadku. Obsługiwany jest również pakiet PyTorch Lightning.
Generowanie wykresów przy użyciu plotly
Plotly to otwarta biblioteka wspierająca tworzenie wykresów. Jupyterlab umożliwia interaktywną współpracę z silnikiem plotly. Bazowy kernel notatnika (Python 3) wspiera plotly bez potrzeby przeprowadzania dodatkowej konfiguracji:
import numpy as np
import scipy.stats as st
import plotly
import plotly.graph_objs as go
from plotly.offline import download_plotlyjs, init_notebook_mode, plot, iplot
init_notebook_mode(connected=True)
print(plotly.__version__)
x = np.arange(1,101)
y = st.norm().rvs(100)
iplot([{"x": x, "y": y}])
Użycie plotly w środowisku wirtualnym Conda wymaga zainstalowania dodatkowych modułów:
conda create --yes --name myenv ipykernel
conda install --name myenv -c plotly plotly chart-studio nbformat
Przykładowe wykorzystanie plotly w powyższym środowisku wirtualnym:
import plotly.graph_objects as go
fig = go.Figure(data=go.Bar(y=[2, 3, 1]))
fig.show()
Uruchomienie pierwszego przykładu wymaga zainstalowania scipy oraz numpy:
conda install --name myenv numpy scipy
Uwaga: obrazy generowane przez plotly są zapisywane w treści notatki co znacząco wpływa na rozmiar tworzonego pliku. Obecnie, rozmiar notatki nie może przekraczać 100 MB, w przypadku przekroczenia limitu otrzymany zostanie błąd o kodzie 413.
Instalacja scikit
Moduł scikit można zainstalować w środowisku Conda następującymi poleceniami:
conda create --yes --name scikit ipykernel python=3.8.6
conda install --name scikit scikit-learn
Uwaga: wersja scikit dostępna przez kanał firmy Intel aktualnie nie działa:
conda install -c intel --name skl scikit-learn # nie zadziała
Biblioteka Tensorflow
Tensorflow to biblioteka wykorzystywana w uczeniu maszynowym i w głębokich sieciach neuronowych. W ramach platformy dostępna jest wersja 2.7.0. Tensorflow jest biblioteką związaną z językiem Python dlatego też, aby móc z niej korzystać z poziomu notatnika istnieje potrzeba ustawienia w prawym górnym rogu odpowiedniego dla tego języka kernela, tak jak jest to zaprezentowane w tym paragrafie.
Przykładowy model sieci neuronowej w ramach biblioteki TensorFlow można zbudować za pomocą wysokopoziomowego interfejsu bazującego na bibliotece Keras:
import tensorflow
from tensorflow import keras
model = keras.Sequential([
keras.layers.Reshape(target_shape=(28 * 28,), input_shape=(28, 28)),
keras.layers.Dense(units=256, activation='relu'),
keras.layers.Dense(units=192, activation='relu'),
keras.layers.Dense(units=128, activation='relu'),
keras.layers.Dense(units=10, activation='softmax')
])
Po stworzeniu modelu należy na podstawie dostępnych danych go wytrenować, a na końcu dokonać predykcji.
model.compile(optimizer='adam',
loss=tf.losses.CategoricalCrossentropy(from_logits=True),
metrics=['accuracy'])
history = model.fit(
train_dataset.repeat(),
epochs=10,
steps_per_epoch=500,
validation_data=val_dataset.repeat(),
validation_steps=2
)
predictions = model.predict(val_dataset)
Tworzenie diagramów - DrawIO
Platforma daje użytkownikowi możliwość tworzenia diagramów (np. w notacji UML) . W celu stworzenia pliku zawierającego diagram trzeba kliknąć na znajdującą się w lewym górnym rogu zakł adkę o nazwie Diagram i wybrać opcję File->Diagram:
Poza tworzeniem pliku dostępne są jeszcze inne polecenia, na przykład związane z edycją pliku.
Na poniższym rysunku został zaprezentowany wygląd panelu służącego do tworzenia diagramów:
Zainstalowane biblioteki
Użytkownicy mają dostęp do wszystkich bibliotek z bazowego obrazu Jupyter oraz następujących bibliotek:
- nb_conda
- ipykernel
- astropy
- plotly
- chart_studio
- ipyfilechooser
- ipyleaflet
- opencv-contrib-python
- lxml
- moviepy
- odfpy
- openpyxl
- statsmodels
- windrose
- ipympl
Pomoc
Jeśli potrzebujesz pomocy napisz do nas na adres: bigdata@task.gda.pl